原核轉錄組知識大放送!一文帶你走進原核轉錄組的世界

同為研究微生物產品的原核轉錄組,不同于擴增子與宏基因組產品。16S/18S/ITS 統稱為擴增子產品,是對特定目標區域進行擴增后再進行測序分析。宏基因組(Metagenome)也稱元基因組,是對微生物整個基因片段進行測序的研究方法。要覆蓋微生物整個基因片段,所以宏基因組比擴增子需要的數據量更多,有更完整的基因序列因此也可以進行基因功能注釋分析。然而,原核轉錄組是一種單菌落微生物轉錄本測序,能夠獲得轉錄本的結構信息及表達信息,基于NGS平臺,通過除核糖體RNA、構建鏈特異性文庫,從基因序列水平和表達水平獲得原核生物在某個時期或者在某種環境條件下轉錄出來的所有轉錄本(包括mRNA,非編碼RNA等)的表達量差異信息及功能特征,找到關鍵的差異功能基因,揭示微生物不同表型形成的分子調控機制。
原核轉錄組結構特征:原核生物是多順反子 mRNA,具有5''、3'' UTR,不具有 poly(A)尾巴,轉錄與翻譯同時進行,壽命期短,具有可變操縱子結構。
1、原核有參轉錄組
對于有參考基因組的物種,我們優先選用原核有參轉錄組流程。分析主要包括數據產出統計、參考基因組比對、基因組裝與結構預測、基因表達水平分析、差異表達基因分析、非依賴 Rho 因子的終止子預測、sRNA 序列預測、SD 序列預測、GSEA、SNP等內容。
2、原核無參轉錄組
對于基因組尚未測序和注釋的生物體,轉錄組需要利用測序數據重新組裝。基于參考的轉錄組組裝和從頭轉錄組組裝都存在許多成熟的計算工具。然而,大多數工具主要是為真核轉錄組設計的,細菌基因組通常比真核基因組更密集,并且相鄰的細菌轉錄本經常重疊,這使得區分相鄰細菌轉錄本的邊界具有難度。多順反子信息使細菌轉錄組組裝進一步復雜化,特別是當在不同條件下使用操縱子的不同啟動子時。此外,真核生物中非編碼RNA的模型通常不適用于細菌中常見的小調節RNA。
Rockhopper 2[1]?軟件結合兩個數據結構,de Bruij 圖和 Burrows-Wheeler 指數,使用類似于 RPKM 的測量來估計轉錄本豐度水平,該測量將轉錄本的讀段數相加,除以轉錄本的長度和歸一化因子。Rockhopper 2 使用更強大的上四分位數轉錄本表達歸一化,使用了高質量的轉錄組組裝,并且在細菌數據組裝上優于其他領先的組裝軟件如:Trinity、SOAPdenovo2。
原核無參轉錄組分析主要包括數據產出統計、轉錄本組裝、基因表達水平分析、差異表達基因分析、GSEA等內容。除轉錄本組裝外,與原核有參轉錄組分析結果基本重疊。
*如果沒有合適參考基因組,也可以搭配做三代測序的細菌小基因組組裝,獲得相對更精確、完整的基因組信息和注釋用于原核轉錄組分析。

實驗提取樣品總 RNA,并進行 RNA 質量檢測,去除核糖體 RNA,在 cDNA 二鏈合成時以 dUTP代替 dTTP,然后連接不同接頭,再利用 UNG 酶法將含有 dUTP 的一條鏈進行消化,只保留連接鏈不同接頭的 cDNA 一鏈,最后進行 PCR 擴增,使用測序儀進行測序。

原核轉錄組實驗流程

原核轉錄組分析流程(有參)
1、參考基因組比對
采用 Rockhopper 2 軟件進行參考基因組比對分析,該軟件采用的比對算法類似 Bowtie2, 基于 BWT(Burrows–Wheeler_transform)數據轉化算法構建參考基因組的 FM-index,使比對更加準確快速。
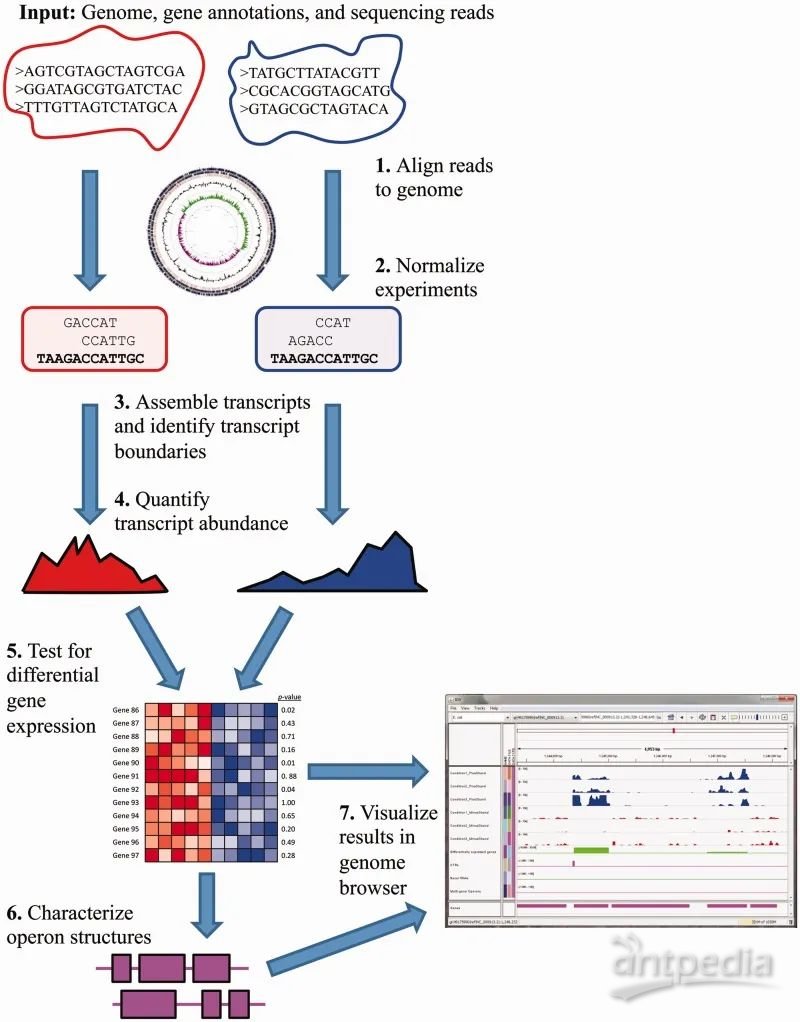
Rockhopper2參考基因組比對分析原理
*對于無參考基因組的細菌微生物,采用 Rockhopper 2軟件 denovo 轉錄組組裝,將測序讀數與轉錄本對齊,從而估計轉錄本豐度水平。

Rockhopper 2 從頭轉錄組序列組裝原理
2、基因表達水平分析
用已知的參考基因序列做為數據庫,采取序列相似性比對的方法鑒定出各基因在各樣本中的表達豐度,使用 Rockhopper 2 軟件獲取每個樣本中比對到基因上的 reads 數以及計算基因的表達量 RPKM 值。RPKM法能消除基因長度和測序量差異對計算基因表達的影響。
PCA 可以從不同維度展現樣本間的關系。樣本聚類距離或者 PCA 距離越近,說明樣本越相似,各組樣本分布在二維或三維空間的不同區域,同組的樣本在空間分布比較集中。
PCA圖
3、差異分析
使用 DEseq2[2] 計算差異基因,其中可視化展示的火山圖可以了解差異表達基因的整體分布情況。
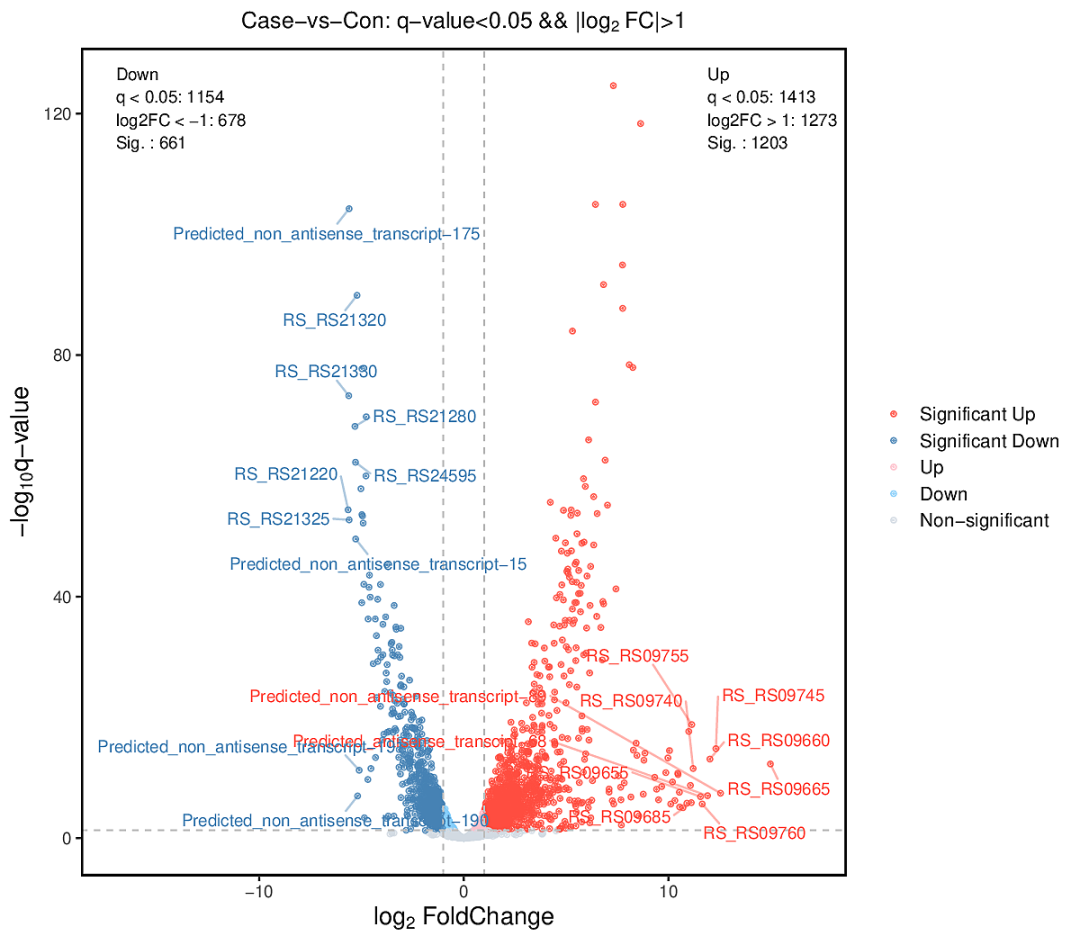
差異基因火山圖
4、富集分析
4.1、GO 富集分析?
GO 富集分析 top30 (篩選三種分類中對應差異基因數目大于 2 的 GO 條目,按照每個條目對應的 -log10pvalue 由大到小排序的各 10 條)條形圖展示如下:

GO富集條形圖
4.2、KEGG 富集分析
KEGG是有關 Pathway 的主要公共數據庫,利用 KEGG 數據庫對差異蛋白編碼基因進行 Pathway 分析(結合 KEGG 注釋結果),并用超幾何分布檢驗的方法計算每個 Pathway 條目中差異基因富集的顯著性。

KEGG富集氣泡圖
4.3 GSEA
基因集富集分析 (Gene Set Enrichment Analysis, GSEA) [3]是一種用于確定一組預先定義的基因是否在兩種生物狀態(例如表型)之間顯示出統計上顯著的或一致的差異的計算方法。其分為三個步驟,分別為計算富集分數、估計富集分數顯著性水平和矯正多重假設驗證。GSEA 分析是基于全部檢出基因進行分析,同時對基因集進行了過濾,默認的標準是基因集最小基因數量為 15、基因集最大基因數量為 500。
?GSEA 示例圖
基因分組聚類示例圖
5、高級分析
5.1、基因組裝與結構預測
用 Rockhopper 2 軟件將測序結果得到基于序列比對數據獲得基因圖譜,將該圖譜與參考基因注釋進行比較,鑒定邊界和新基因。
統計文件包括預測非反義轉錄本數目、預測轉錄本數目、預測多基因操縱子數目、預測反義 RNAs 數目、差異表達的蛋白編碼基因數目、5'' 端 UTR 數目、3'' 端 UTR 數目。
根據鑒定轉錄起始位點和轉錄終止位點以及注釋文件中的翻譯起始位點和翻譯終止位點預測得到 UTR 位置信息及其長度信息:
3'' 端UTR長度統計圖
5'' 端UTR長度統計圖
5.2、操縱子預測
原核生物功能上相關的幾個基因往往串聯在排列在一起,構成操縱子結構作為一個表達單位,用Rockhopper 2 軟件將操縱子預測算法從純序列特征發展到結合測序實驗數據(即計算所得基因表達量),即聯合基因間距離和基因表達量相關性兩個特征用樸素貝葉斯分類器模型來預測操縱子。對預測得到操縱子進行長度分布、包含的結構基因數目和操縱子鏈分布進行計算和可視化。
操縱子長度分布圖
操縱子結構基因數目統計圖
操縱子鏈分布圖
5.3、反義基因預測
新預測的基因中如果基因與已知編碼基因重疊或包含,且位于不同的鏈上,則該基因判定為反義基因,使用 Rockhopper 2 軟件預測,反義基因分為三種類型:全部包含(enclosed),3'' 端重疊(convergent)和5'' 端重疊(divergent)。在測序數據來源于鏈特異文庫的條件下,可以預測反義基因位置、類型和數量。
反義基因統計圖
5.4、非依賴 Rho 因子的終止子預測
原核生物基因組中有轉錄終止信號,稱為終止子,部分基因轉錄終止需要輔助蛋白 Rho 因子,但其它基因核心酶本身即可終止轉錄。不依賴于 Rho 因子的轉錄終止子具有兩個重要結構特征:DNA 順序有雙重對稱(dyad),位于 RNA 3'' 端之前 15-20 核苷酸處和 DNA 模板鏈中有一串約 6 個 A,轉錄為 RNA ?3'' 端的U。雙重對稱的意義在于其基因能形成發夾結構。采用 TransTermHP[4] 軟件預測不依賴于 Rho 因子的終止子序列。
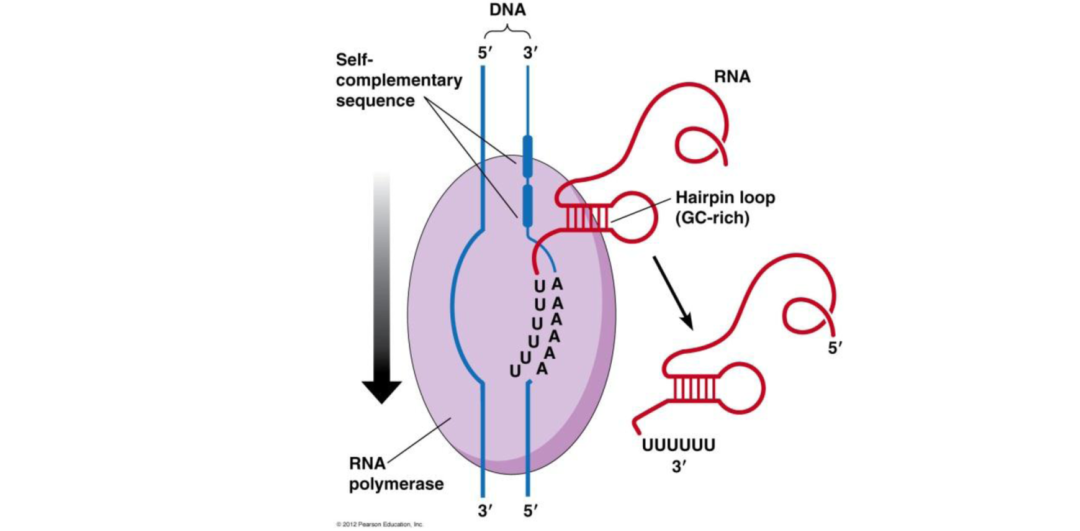
轉錄終止子示意圖
5.5、sRNA 序列預測
原核生物 sRNA 是一類長度在 50-500 bp 的小 RNA 分子,用 Rockhopper 2 軟件預測 Novel 基因,RNAFold 分析其莖環結構,進行二級結構預測,使用 IntaRNA[5] 進行靶基因預測,可綜合判斷 Novel 基因是否為潛在的 sRNA。
5.6、SD 序列預測
SD(Shine-Dalgarno)序列僅存在于原核生物中,SD 序列是一個存在于信使 RNA 上的核糖體結合位點,通常位于起始密碼子上游。除引導翻譯過程外 SD 序列還有調控翻譯效率的作用。采用 RBSfinder[6] 軟件預測包含 SD 序列。
SD 序列示意圖
5.7、SNP 分析
SNP(Single Nucleotide Polymorphisms,單核苷酸多態性),是指在基因組上單個核苷酸的變異,包括置換、顛換、缺失和插入。以組裝好的轉錄本為模板序列,將原始序列與其進行比對,利用 samtools 軟件進行染色體坐標排序、去重等處理,再用 samtools、bedtools 等軟件預測樣本中的 SNP 和 INDEL 位點。然后利用 snpEff 等軟件進行功能注釋。為了降低 SNP&INDEL 檢測的錯誤率,使用 QUAL (A quality score associated with the inference of the given allele) 大于等于 20,且 DP(combined depth across samples)大于等于 4 進行過濾結果。對 SNP/INDEl 在基因組上各功能區域的分布進行統計。
01
通過工程改造的冷營養 將半纖維素有效轉化為 2, 3-丁二醇:機制和效率
Efficient conversion of hemicellulose into 2, 3-butanediol by engineered: mechanism and efficiency
發表期刊:Bioresource Technology
影響因子:11.889
文章鏈接:https://www.sciencedirect.com/science/article/abs/pii/S0960852422007829
02
AI-2/LuxS系統在冷藏鮮蝦生物保存中的作用:增強植物乳桿菌對營養物質的競爭能力
The role of AI-2/LuxS system in biopreservation of fresh refrigerated shrimp: Enhancement in competitiveness of Lactiplantibacillus plantarum for nutrients
發表期刊:Food Research International
影響因子:7.425
文章鏈接:https://www.sciencedirect.com/science/article/abs/pii/S0963996922008961
03
蒙脫石對鎘誘導大腸桿菌耐藥基因的調控機制
Regulatory mechanism of montmorillonite on antibiotic resistance genes in Escherichia coli induced by cadmium
發表期刊:Applied Microbiology and Biotechnology
影響因子:5.56
文章鏈接:https://link.springer.com/article/10.1007/s00253-022-12075-x
04
比較轉錄組結合形態生理學分析揭示了嗜熱四膜蟲捕食誘導嗜水氣單胞菌抗噬菌體防御的分子機制
Comparative transcriptome combined with morphophysiological analyses revealed the molecular mechanism underlying Tetrahymena thermophila predation-induced antiphage defense in Aeromonas hydrophila
發表期刊:Virulence
影響因子:5.428
文章鏈接:https://www.ncbi.nlm.nih.gov/pmc/articles/PMC9518995
以上就是原核轉錄組的相關介紹內容,感興趣的老師可以聯系我們做進一步詳細溝通。
參考文獻
[1].De novo assembly of bacterial transcriptomes from RNA-seq data. Brian Tjaden. Genome Biology, 16:1, 2015
[2].Love M I , Huber W , Anders S . Moderated estimation of fold change and dispersion for RNA-seq data with DESeq2[J]. Genome Biology, 2014.?
[3].Aravind Subramanian, Pablo Tamayo, Vamsi K. Mootha. Gene set enrichment analysis: A knowledge-based approach for interpreting genome-wide expression profiles[J]. PNAS, 2005
[4].C. Kingsford, K. Ayanbule and S.L. Salzberg. Rapid, accurate, computational discovery of Rho-independent transcription terminators illuminates their relationship to DNA uptake[J]. Genome Biology, 2007
[5].Martin Mann, Patrick R. Wright, and Rolf Backofen. IntaRNA 2.0: enhanced and customizable prediction of RNA-RNA interactions[J]. Nucleic Acids Research, 2017
[6].Chang TH, Huang HY, Hsu JB, Weng SL, Horng JT, Huang HD. An enhanced computational platform for investigating the roles of regulatory RNA and for identifying functional RNA motifs[J]. BMC Bioinformatics, 2013
上海歐易生物醫學科技有限公司(簡稱:“歐易生物”),成立于2009年,經過十多年穩健發展,已經成長為擁有“晶準生物”“鹿明生物”“青島歐易”三家全資子公司,近600名員工的生物科技領域集團型企業。
歐易生物始終秉持著“硬數據 · 好服務”的理念服務于大眾。為大生命科學、大健康相關研究領域,以及醫藥、食品及日化企業的客戶,提供從基礎研究到藥物靶點發現、藥理藥效及安全性評價、疾病分子標志物篩選、致病菌及耐藥菌溯源等相關技術服務,全力加速客戶研究與開發進程,提升客戶研究與開發價值。

歐易生物攜手旗下子公司,實現了中心法則上、中、下游多層組學的串聯,從基因組、轉錄組、表觀組、微生物組,到蛋白組、代謝組及近年熱門的單細胞&空間多組學技術服務,為科研用戶提供全面的創新多組學技術服務。

歐易生物已先后獲得上海市科技小巨人企業、閔行區研發機構、閔行區企業技術中心、產權管理體系認證企業等資質。擁有授權發明專利30+項,在受理發明專利50+項,軟件著作權150+項。